测序文件原始数据下载
fastq,SRR等数据下载
1. 背景
美国国立生物技术信息中心(National Center for BiotechnologyInformation),即我们所熟知的NCBI是由美国国立卫生研究院(NIH)于1988年创办。创办NCBI的初衷是为了给分子生物学家提供一个信息储存和处理的系统。
除了建有GenBank核酸序列数据库(该数据库的数据资源来自全球几大DNA数据库,其中包括日本DNA数据库DDBJ、欧洲分子生物学实验室数据库EMBL以及其它几个知名科研机构)之外,NCBI还可以提供众多功能强大的数据检索与分析工具。目前,NCBI提供的资源有Entrez、Entrez Programming Utilities、MyNCBI、PubMed、PubMed Central、EntrezGene、NCBI Taxonomy Browser、BLAST、BLAST Link (BLink)、ElectronicPCR等共计36种功能.
上述的界面主要包含下面的部分
- 下拉列表,可以查询相对应的数据库
- 输入关键词进行相关检索
- 资源库导航栏,位于左侧未知,点击后可查看资源库涵盖的数据库
- 数据提交、下载、分析工具和帮助指南等
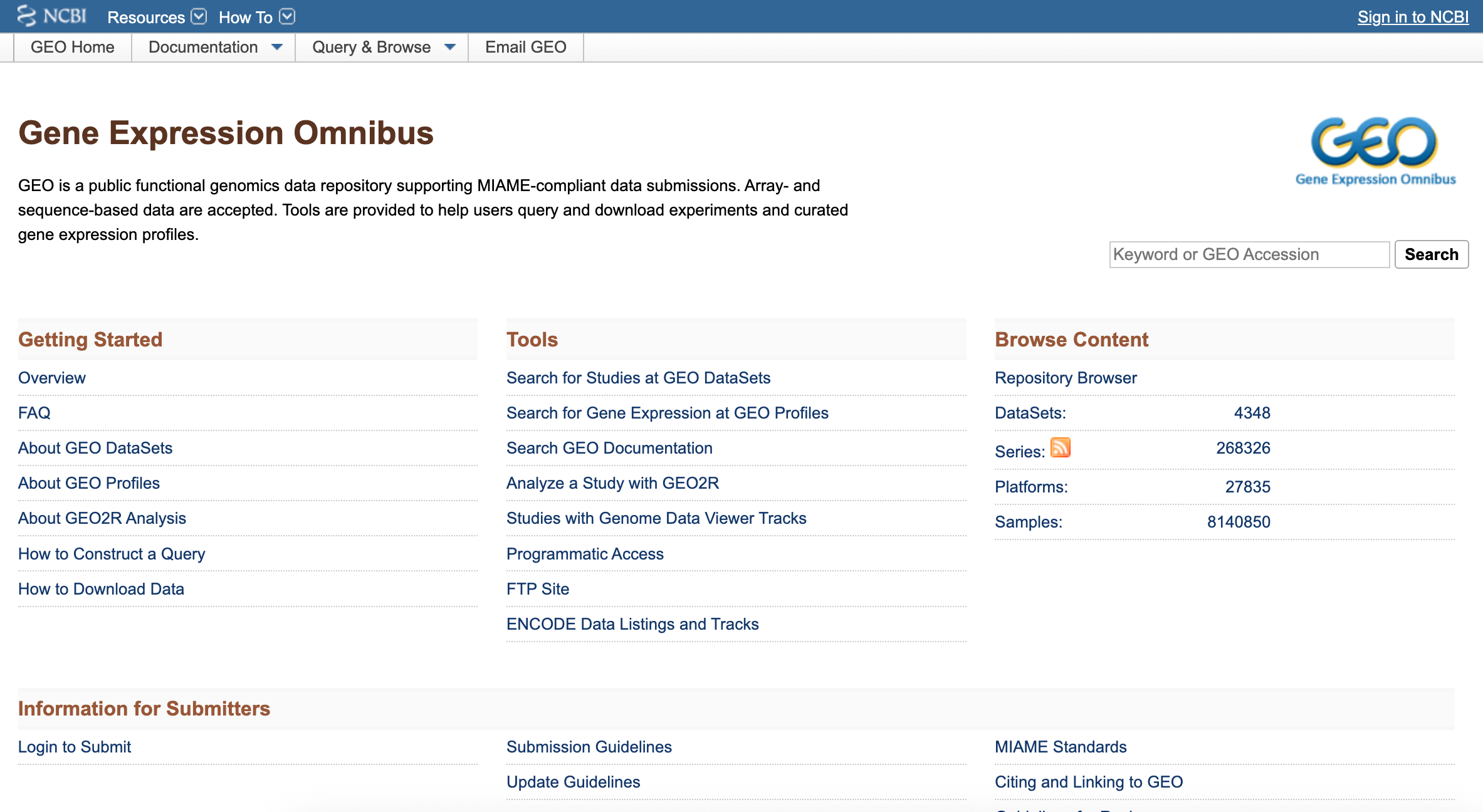
1.1 GEO数据库
GEO数据库全称Gene Expression Omnibus database,是由美国国立生物技术信息中心NCBI创建并维护的基因表达数据库。
它创建于2000年,收录了世界各国研究机构提交的高通量基因表达数据,也就是说只要是目前已经发表的论文,论文中涉及到的基因表达检测的数据都可以通过这个数据库中找到。
数据库中的数据都是免费的!
1.2 GEO数据类型
在下载数据之前需要了解GEO数据库的存放类型
- GSE数据编号(Series)
- GPL数据编号(GEO platforms)
- GSM数据编号(Samples)
- GDS数据编号(Datasets)
一篇文章可以有一个或者多个GSE(Series)数据集
一个GSE里面可以有一个或者多个GSM(Samples)样本,而每个数据集都有着自己对应的芯片平台,就是GPL(GEO platforms)。
GSE编号一般为作者提交时生成的原始数据编号,后续NCBI中的工作人员会根据研究目的、样品类型等信息归纳整合为一个GDS(Datasets),整理后的数据还会有GEO profile数据
2. ASCP下载
ASCP是用来大数据下载的神器,但是无法下载,下面记录了一部分,等解决了再补充
如果已经获取到 SRR 号,我们还可以通过脚本直接下载对应的数据,无需访问 EBI 网站以获取链接。
ascp -v -QT -l 300m -P33001 -k 1 -i /home/erwin/micromamba/envs/SC/etc/asperaweb_id_dsa.openssh era-fasp@fasp.sra.ebi.ac.uk:/vol1/fastq/SRR113/062/SRR11396162/SRR11396162_1.fastq.gz .2.1 问题排查
- 问题排除
将命令行输入到bash之后,不报错也不进行下载
- 第一步
先确定是否网址是正确的
curl -I https://ftp.sra.ebi.ac.uk/vol1/fastq/SRR113/062/SRR11396162/SRR11396162_1.fastq.gz返回的结果是
HTTP/1.1 200 OK
Date: Fri, 05 Dec 2025 14:06:36 GMT
Server: Apache
Last-Modified: Sat, 27 Jun 2020 20:05:36 GMT
ETag: "1434079ea-5a916594ac400"
Accept-Ranges: bytes
Content-Length: 5423266282
Access-Control-Allow-Origin: *
Access-Control-Allow-Methods: GET,OPTIONS
Access-Control-Allow-Headers: Authorization,Origin,User-Agent,X-Requested-With,If-Modified-Since,Cache-Control,Content-Type,Range,Accept Content-Type: application/x-gzip- 第二步
ascp不下载,不是文件或路径问题,基本上是处在Aspera通道本身
nc -zv fasp.sra.ebi.ac.uk 33001Connection to fasp.sra.ebi.ac.uk 33001 port [tcp/*] succeeded! 提示连接成功
- 第三步
修改文件之后也无法进行下载,可能ascp已经被放弃使用了,考虑别的方向
2.2 ASCP更新4.20.0
将ascp更新到4.20.0进行尝试
- 第一步 创建全新的环境之后进行安装,避免不必要的冲突
#安装asper
mamba install -c conda-forge -c bioconda asper-cli=4.20.0
# 寻找路径
which ascli- 第二步
下载sdk
# 下载
wget https://production-transfer-sdk.s3.us-west-2.amazonaws.com/1.1.2/linux-amd64-1.1.2-753f662.tar.gz
# 解压
tar -zxvf linux-amd64-1.1.2-753f662.tar.gz
# 安装 ascp,注意:命令需在与linux-amd64-1.1.2-753f662.tar.gz文件同级目录下运行
ascli config ascp install --sdk-url=file:///linux-amd64-1.1.2-753f662.tar.gz
# 寻找 ascp 路径
ascli config ascp show
# /path/to/your/ascp- 第三步
寻找密钥
在下载的路径中寻找新版的密钥
# 找到秘钥,主要使用pem
find $HOME -name aspera_bypass_dsa.pem
# 下载
ascp -QT -l 300m -P33001 -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh anonftp@ftp.ncbi.nlm.nih.gov:/sra/sra instant/reads/ByRun/sra/SRR/SRR123/SRR1234567/SRR1234567.sra .最终找不到密钥更换本地下载,有博文说可以更换原来的密钥,但是尝试了也无反应
2.3 本地安装
aspera-cli 是一个由 Ruby 编写的命令行工具,它提供了一个统一的接口 (ascli) 来与 IBM Aspera 传输平台进行交互。
ascli 和 ascp 之间的关系是 上层工具和底层核心协议执行程序 的关系。简单来说,所有的 Aspera 高速文件传输最终都是由 ascp(或其继任者 ascp4)执行的,而 ascli 是一个更高级、更智能的命令行前端,用于简化和自动化复杂的传输任务,尤其是与 Aspera 平台产品(如 Aspera on Cloud, Faspex 等)交互时。
按照ascli的教程安装
- 第一步
更换镜像源,国外连接的太慢了
gem sources --remove https://rubygems.org/
# 更换china
gem sources -a https://gems.ruby-china.com/
# 检验
gem sources -l
# 安装
sudo gem install aspera-cli网络限制导致Ruby 无法下载和更新,新版的ascp-cli需要Ruby > 3.1,原始安装的版本太低,最终放弃此方法
3. Kingfisher下载
kingfisher是一款专门用于高通量测序数据下载的工具。支持从公共数据库(ENA、NCBI、SRA、Amazon AWS 和 Google Cloud)获取序列文件及其元数据注释。
- 第一步.
使用conda 或者mamba 进行kingfisher进行
mamba install -c conda-forge -c bioconda kingfisher- 第二步.
选择合适的平台去合理的下载
| Method | 描述 (Description) | 速度 (Speed) | 格式 (Format) | 工具依赖 (Tool Dependency) | 账户/费用 (Account/Cost) |
|---|---|---|---|---|---|
| ena-ascp | 通过 Aspera (FASP) 协议从 ENA 下载预先生成的 .fastq.gz 文件 | 最快(利用 Aspera 高速传输) | .fastq.gz | aspera-cli (ascp) | 无 |
| ena-ftp | 通过 cURL 或类似工具从 ENA 下载预先生成的 .fastq.gz 文件 | 较快(无需 fasterq-dump) | .fastq.gz | curl 或 wget | 无 |
| prefetch | 使用 NCBI 的 prefetch 下载 .SRA 文件,然后用 fasterq-dump 提取 FASTQ | 中等(需额外 fasterq-dump 步骤) | .SRA | prefetch, fasterq-dump | 无 |
| aws-http | 使用 aria2c (多线程 HTTP) 从 AWS Open Data 下载 .SRA 文件,然后用 fasterq-dump 提取 | 中等(需额外 fasterq-dump 步骤) | .SRA | aria2c, fasterq-dump | 通常无 |
| aws-cp | 使用 aws s3 cp 从 AWS Open Data 下载 .SRA 文件,然后用 fasterq-dump 提取 | 中等(需额外 fasterq-dump 步骤) | .SRA | aws-cli, fasterq-dump | 通常不需要支付费用或 AWS 账户 |
| gcp-cp | 使用 Google Cloud gsutil 下载 .SRA 文件,然后用 fasterq-dump 提取 | 中等(需额外 fasterq-dump 步骤) | .SRA | gsutil, fasterq-dump | 需要支付费用和 Google Cloud 账户 |
# 下载整个bioproject
kingfisher get -p PRJNA614539 -m ena-ascp ena-ftp prefetch aws-http 1>download.log 2>&1
# 下载单个样本
kingfisher get -r SRR11396159 -m prefetch aws-http
# 下载多个样本
kingfisher get --run-identifiers-list SRR_list.csv -m ena-ascp ena-ftp prefetch --download-threads 10 --check-md5sums 1>down_srr_list.log 2>&1- -p :批量下载BioProject IDs 中的所有数据
- -m :指定下载源 ena-ascp、ena-ftp、prefetch、aws-http、aws-cp、gcp-cp 等
- -r :下载某个确定的SRA数据
- –run-identifiers-list :SRR样本列表文件,单列SRR号
- –download-threads -t : 指定线程数
3.1 Kingfisher使用
- 使用
annoteat查询SRR数据.
# 全部信息
kingfisher annotate -r SRR11396159 --all-column -f tsv -o ./SRR11396159.srr.list
# 简要信息
kingfisher annotate -r SRR11396159 -f tsv -o ./SRR11396159.srr.list- 使用
get进行下载
- ena-asc 通过Aspera从ENA下载.fastq.gz文件
- ena-ftp 通过curl从ENA下载.fastq.gz文件
- prefetch 使用NCBI的prefetch从sra-tools下载.SRA文件,然后用fasterq-dump提取
- aws-http 使用aria2c通过多个连接线程从AWS Open Data Program下载.SRA文件,之后用fasterq-dump提取
# 单个样本 ftp调用arias2
kingfisher get -r SRR11396174 -m ena-ftp --download-threads 8 --check-md5
# 单个样本 调用ascp
kingfisher get -r SRR11396174 -m ena-ascp --download-threads 5ena-ftp 非常容易ftp被ENA拒绝,然后断开下载,甚至无法恢复
Kingfisher更像是一个整合的工具,其实就是调用ascp,prefetch,aria2c等工具进行相关的下载,但是因为是整合工具所以自定义的性能自然变差,最后还是要回归到原始的下载工具,小的文件是可以使用的,非常方便。
4. aria2下载
Aria2是一款轻量级、多协议、跨平台命令行下载工具,支持HTTP/HTTPS、FTP、BitTorrent和Metalink等协议。它通过多线程、多源下载来提高下载速度,占用资源少,且支持多种操作系统,包括Windows、macOS、Linux等。
aria2 是一款轻量且高效命令行下载工具,它提供了对多协议和多源地址的支持,并尝试将下载带宽利用率最大化,目前支持的协议包括HTTP(S)、FTP、BitTorrent(DHT, PEX, MSE/PE) 和 Metalink。通过 Metalink 的分块检查,aria2 可以在下载过程中自动的进行数据校验。
4.1 aria2安装
Mac安装。
# Mac
brew install aria2
# Ubuntu
sudo apt-install aria24.2 aria2使用
aria2本身没有问题,主要是下载来源的封禁
aria2c -x 8 -s 8 -c -m 5 --retry-wait 10 ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR113/059/SRR11396159/SRR11396159_1.fastq.gz- x 8个线程同时下载。
- s 分为8段下载
- c 断点下载
- m 试探下载5次
- -retry-wait 10 等待10秒
下载开始后,会形成 SRR11396173_1.fastq.gz.aria2 是aria2的下载控制/临时文件,用来记录当前文件的下载速度和状态,支持断点续传
- 已经下载了多少字节
- 文件被分成了多少块
- 每块下载到哪
- 校验信息(hash)
- 下载 URL / 状态信息
4.3 自动重新下载
- 第一步
在ENA中得到ftp的文件,开始进行信息提取与筛选
使用awk 进行文本的操作~
awk -F'\t' 'NR>1{
n=split($3,a,";"); # 按分号拆分每行的 fastq_ftp
for(i=1;i<=n;i++) # 遍历每个文件
print "ftp://" a[i] # 输出并加 ftp:// 前缀
}' aria2.txt > links.txt将文件中的下载地址全部按行排列,排列好的下载链接用来做循环
- 第二步
写一个脚本,因为aria2c经常被ENA发送EOF拒绝,然后导致下载结束
写个循环强制进行重新下载,直到下载成功为止!!!
为了防止文件的冲突,添加了将下载好的文件移动到文件夹中
#!/bin/bash
# --- 脚本配置 ---
# 包含所有 FTP/HTTPS 链接的文件路径
LINKS_FILE="links.txt"
# 目标存放文件
TARGET_DIR="dataSRR"
# 文件夹不存在创建
mkdir -p "$TARGET_DIR"
# aria2c 下载参数
# -c: 启用断点续传(必须)
# -x 5 -s 5: 并发连接数和分块数(推荐值,您可以根据网络情况调整)
# --retry-wait 60: 失败后等待 60 秒再重试
# --max-tries 0: 设置为 0,让 aria2c 内部无限重试
ARIA2_OPTIONS="-c -x 5 -s 5 --retry-wait 15"
# 如果下载彻底失败(达到 aria2c 的最大重试次数后退出),外部脚本等待的秒数
# 只有当 aria2c 内部重试机制失效时,外部脚本才会在等待后进行下一次尝试
EXTERNAL_SLEEP_SECONDS=15
echo "--- 批处理下载任务开始 ---"
echo "链接文件: ${LINKS_FILE}"
echo "aria2c 参数: ${ARIA2_OPTIONS}"
echo "--------------------------"
# 检查链接文件是否存在
if [ ! -f "$LINKS_FILE" ]; then
echo "❌ 错误: 链接文件 ${LINKS_FILE} 不存在。"
exit 1
fi
# 1. 逐行读取 links.txt 文件中的链接
while IFS= read -r DOWNLOAD_URL; do
# 清理行首尾空格,并跳过空行
DOWNLOAD_URL=$(echo "$DOWNLOAD_URL" | xargs)
if [ -z "$DOWNLOAD_URL" ]; then
continue
fi
FILE_NAME=$(basename "$DOWNLOAD_URL")
echo ""
echo "=========================================================="
echo "🚀 正在处理文件: ${FILE_NAME}"
echo "=========================================================="
# 2. 无限循环:直到下载成功为止
while true; do
echo "$(date): 尝试下载/续传..."
# 运行 aria2c
aria2c ${ARIA2_OPTIONS} "$DOWNLOAD_URL"
# 检查 aria2c 的退出状态码
EXIT_CODE=$?
if [ ${EXIT_CODE} -eq 0 ]; then
# 退出码为 0,表示下载成功
echo "✅ $(date): 文件 ${FILE_NAME} 下载成功!"
mv -v "$FILE_NAME" "$TARGET_DIR"
break # 跳出内部循环,处理下一个链接
else
# 退出码不为 0,表示彻底失败(达到内部最大重试次数)
echo "❌ $(date): 下载失败 (退出码: ${EXIT_CODE}),等待 ${EXTERNAL_SLEEP_SECONDS} 秒后重试..."
sleep ${EXTERNAL_SLEEP_SECONDS}
# 脚本会再次进入循环,重新执行 aria2c 进行续传
fi
done
done < "$LINKS_FILE"
echo ""
echo "--- 批处理下载任务全部完成! ---"5. md5sum检验文件
使用bash脚本进行文件的检验,核心就是对比提供的MD5和文件校验的MD5比较
自己设置的话主要是文件的命名和位置
还有就是md5的文件格式,内容也要注意,核心就是提取每个文件的md5进行比较~
#!/bin/bash
# ======================================
# 🚀 批量 FASTQ 文件 MD5 校验脚本
# ======================================
# --- 配置 ---
# 请将你的元数据文件命名为 md5sum.txt,或修改这里的路径
METADATA_FILE="md5sum.txt"
# --- 配置结束 ---
echo "--- 🚀 开始批量 FASTQ MD5 校验 ---"
echo "--- 校验结果: [文件名称] [校验结果] ---"
# 检查元数据文件是否存在
if [ ! -f "$METADATA_FILE" ]; then
echo "错误: 元数据文件 '$METADATA_FILE' 未找到。请检查文件名或路径。"
exit 1
fi
# 使用 awk 解析元数据并进行 MD5 校验
awk '
{
# 跳过空行或注释行
if ($1 == "" || $1 ~ /^#/ || $1 == "run_accession") next
# 提取 SRR ID 和预期的 MD5 校验码
srr_id = $1
split($2, expected_md5s, ";") # 以分号分隔 MD5
# 构造本地文件名
file1_name = srr_id "_1.fastq.gz"
file2_name = srr_id "_2.fastq.gz"
# ---------- 校验文件 1 ----------
if (system("test -f " file1_name) != 0) {
print file1_name, "FILE_MISSING"
} else {
# 计算本地文件的 MD5
cmd = "md5sum " file1_name
cmd | getline calculated_md5_output
close(cmd)
split(calculated_md5_output, calculated_md5_array, " ")
calculated_md5 = calculated_md5_array[1]
# 比较 MD5
expected_md5 = expected_md5s[1]
if (calculated_md5 == expected_md5) {
print file1_name, "OK"
} else {
print file1_name, "FAILED!"
print " 预设值: " expected_md5
print " 计算值: " calculated_md5
}
}
# ---------- 校验文件 2 ----------
if (system("test -f " file2_name) != 0) {
print file2_name, "FILE_MISSING"
} else {
# 计算本地文件的 MD5
cmd = "md5sum " file2_name
cmd | getline calculated_md5_output
close(cmd)
split(calculated_md5_output, calculated_md5_array, " ")
calculated_md5 = calculated_md5_array[1]
# 比较 MD5
expected_md5 = expected_md5s[2]
if (calculated_md5 == expected_md5) {
print file2_name, "OK"
} else {
print file2_name, "FAILED!"
print " 预设值: " expected_md5
print " 计算值: " calculated_md5
}
}
}
' "$METADATA_FILE"
echo -e "\n--- ✅ MD5 校验完成 ---"