CellRanger快速使用
Cellranger分析10x单细胞数据
1. 背景
在单细胞基因组学领域,CellRanger 是10x Genomics公司推出的一款核心分析工具,它专门设计用于处理和分析来自10X Genomics平台的单细胞测序数据。CellRanger工具的设计理念在于提供一个端到端的解决方案,从原始数据的预处理到最终的基因表达矩阵生成。这个工具集成了数据拆分、基因表达定量、聚类分析等多个步骤,极大地简化了单细胞数据的分析流程。
CellRanger 能够高效处理大规模的单细胞测序数据,并且是专门为10X Genomics平台设计的。这意味着它能够充分利用该平台的UMI(Unique Molecular Identifiers)技术,提高数据的准确性和可靠性。UMI技术允许在测序过程中识别和校正错误,这对于单细胞分析尤其重要
Cell Ranger is a set of analysis pipelines for processing Chromium single cell data. It performs barcode processing and UMI counting to quantify gene expression (from 3’, 5’, and Flex assays), assembles V(D)J immune receptor sequences, and analyzes Feature Barcode data for applications such as cell surface protein analysis and sample multiplexing
CellRanger 主要功能:
- Alignment(比对): 将测序数据与参考基因组进行比对
- Clustering(聚类): 将相似的细胞聚集在一起,以便进行进一步分析
- Differential Expression(差异表达):识别在不同条件下表达水平有显著差异的基因
- Dimensionality Reduction(降维):使用技术如PCA(主成分分析)简化数据,使其更易于可视化和分析
- Interactive(交互式):提供交互式工具,以便研究人员可以探索和分析数据
- Marker Genes(标记基因):识别特定细胞类型的标记基因
- Quality Control(质量控制):检查数据质量,去除低质量的细胞
- Quantification(定量):计算每个细胞的基因表达水平
- UMIs(唯一分子标识符):利用UMI技术提高数据准确性
- Visualization(可视化):提供数据可视化工具,帮助理解复杂的数据集
2. CellRanger安装准备
现在写的时候,是出现的Cell Ranger 10.0.0(Nov 13, 205) 安装之前需要进行一系列的检查,cellranger需要比较大的计算资源
- 8 核 Intel 或 AMD 处理器(推荐 64 核),支持 AVX+ 指令集,该指令集于 2011 年在 Intel 和 AMD CPU 上推出。未来需要 AVX2 指令集,该指令集于 2013 年在 Intel CPU 上推出,2015 年在 AMD CPU 上推出
- 64GB RAM内存(128GB 推荐)
- 1.5TB 硬盘
- 64-bit CentOS/RedHat 8.0 or Ubuntu 20.04
| 配置项 | Small | Medium | Large |
|---|---|---|---|
| Dataset details | 20k (3’/5’/Flex - GEX), 10–20k reads/cell | 320k (Flex - GEX+AB), 200 features, 10k reads/cell | 1M (Flex v2 - GEX+AB+CRISPR), 500 features, 10k GEX reads/cell |
| Processor | 8-core Intel or AMD processor, with AVX+ instruction set | 16-core Intel or AMD processor, with AVX+ instruction set | 32-core Intel or AMD processor, with AVX+ instruction set |
| RAM | 64 GB RAM | 256 GB RAM | 512 GB RAM |
| Free disk space | 1.5 TB | 2 TB | 6 TB |
2.1 下载Cell Ranger
第一步 下载Cell Ranger
主要是针对Linux系统,主要是文件格式不同
tar.gz
# curl
curl -o cellranger-10.0.0.tar.gz "https://cf.10xgenomics.com/releases/cell-exp/cellranger-10.0.0.tar.gz?Expires=1765244249&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA&Signature=CGL6ot9P5vJfuQmrIxrt3CXlcPjfA96mae9yRnG4GN-dZrgf4vRTPMZyZ~-7L5AAiNYb84zoNTxDTH~onQo~8P1LfAaqBmJzXv11svx7GpcA7fJN0ihiAs1YvInN2byeZxmweoctL4RLc4DsY1ikobyjuJ2cXVzo6mCaOF84Jv9acgLtptPnO8EGUKp2SNpAUqWw0eDJ95X4R0hNssnJb7s55wiPq3bsD4-UiCOP-Cz9R1JU39Pqh9rX~fha9huGRr83LNZN7PyHiDOto1EdjfrUEzDJTLK9kicOcrtHT8ED5lU4dxLYBy902LdVQmaRDsKBWQufaFkmAqg7f0TMiQ__"
# wget
wget -O cellranger-10.0.0.tar.gz "https://cf.10xgenomics.com/releases/cell-exp/cellranger-10.0.0.tar.gz?Expires=1765244249&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA&Signature=CGL6ot9P5vJfuQmrIxrt3CXlcPjfA96mae9yRnG4GN-dZrgf4vRTPMZyZ~-7L5AAiNYb84zoNTxDTH~onQo~8P1LfAaqBmJzXv11svx7GpcA7fJN0ihiAs1YvInN2byeZxmweoctL4RLc4DsY1ikobyjuJ2cXVzo6mCaOF84Jv9acgLtptPnO8EGUKp2SNpAUqWw0eDJ95X4R0hNssnJb7s55wiPq3bsD4-UiCOP-Cz9R1JU39Pqh9rX~fha9huGRr83LNZN7PyHiDOto1EdjfrUEzDJTLK9kicOcrtHT8ED5lU4dxLYBy902LdVQmaRDsKBWQufaFkmAqg7f0TMiQ__"Cell Ranger v10 highlights - Cell Ranger v10 现在支持 GEM-X Flex v2 数据的分析!用户现在可以在单个 GEM 孔中同时分析多达 384 个样本 - Improvements have been made to the cell calling algorithm.
采用了8.0.0的版本进行分析,数据较老,版本按照自己需求解决
第二步 下载References
基于 Ensembl v110 注释的人类和小鼠参考转录组新版本现已开放下载。详情请参阅参考转录组发布说明。
Human reference (GRCh38) - 2024-A
curl -O "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38-2024-A.tar.gz"
# md5sum: a7b5b7ceefe10e435719edc1a8b8b2fa
# File size:11GBMouse reference (GRCm39) - 2024-A - Mouse reference (GRCm39) dataset required for Cell Ranger
curl -O "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCm39-2024-A.tar.gz"
# File size:9.7GB
#md5sum: 37c51137ccaeabd4d151f80dc86ce0b3Rat reference (mRatBN7.2) - 2024-A 大鼠的参考基因组
curl -O "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-mRatBN7-2-2024-A.tar.gz"
# File size: 9.5 GB
# md5sum: 34d4af0874a45dfa03436efe4eaec2feHuman (GRCh38) and mouse (GRCm39) reference - 2024-A 混合参考基因组
curl -O "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38_and_GRCm39-2024-A.tar.gz"
# File size: 11 GB
# md5sum: b2d628e6c6813e1396989a9a9ca2ef23Human V(D)J reference (GRCh38) - Human reference (GRCh38/Ensembl/10x) dataset required for Cell Ranger V(D)J.
curl -O "https://cf.10xgenomics.com/supp/cell-vdj refdata-cellranger-vdj-GRCh38-alts-ensembl-7.1.0.tar.gz"
# File size: 3.3 MB
# md5sum: 65b5b033723b07fc1bb5375e5645761cMouse V(D)J reference (GRCm38)) - Mouse reference (GRCm38/Ensembl/10x) dataset required for Cell Ranger V(D)J.
curl -O "https://cf.10xgenomics.com/supp/cell-vdj/refdata-cellranger-vdj-GRCm38-alts-ensembl-7.0.0.tar.gz"3. CellRanger 安装
Cell Ranger 以单个独立的 tar 文件形式提供,可解压到系统上的任何位置。它捆绑了所有必需的软件依赖项,这些依赖项已预编译,可在多种 Linux 发行版上运行。为方便起见,Cell Ranger 所需的参考数据包可单独下载。
按照上面的步骤下载好tar版软件之后进行解压
第一步 解压软件
cd /opt
# [ download file from downloads page ] 自定义的文件夹
tar -xzvf cellranger-x.y.z.tar.gz
# 或者解压xz
tar -xvf cellranger-x.y.z.tar.xz第二步 解压参考基因组
# [ download file from downloads page ]
# Example human reference transcriptome
tar -xzvf refdata-gex-GRCh38-2020-A.tar.gz第三步 将cellranger加入到路径中
有临时加入路径或长期加入路径
export PATH=/opt/cellranger-x.y.z:$PATH第四步 测试cellranger
cellranger
# test
cellranger testrun --id=check_install等待测试后,提醒
Waiting 6 seconds for UI to do final refresh.
Pipestance completed successfully!
2022-01-04 12:51:02 Shutting down.
4. CellRnager 使用
Cell Ranger 是一款用于处理 10x Genomics Chromium 仪器生成的单细胞数据集的软件包。Cell Ranger 包含六个主要流程,用于比对 reads、生成特征条形码矩阵、执行聚类和其他辅助分析等等。
cellranger multi推荐用于分析几乎所有 10x Genomics Chromium 单细胞数据集的流程。它输入来自单个 GEM Well 生成的多个文库组合的 FASTQ 文件。Cellranger Multi 可执行比对、过滤、条形码计数和 UMI 计数。它利用细胞条形码生成特征-条形码矩阵,确定细胞簇,进行基因表达分析,并提供初步的细胞类型注释。cellranger count它读取FASTQ文件并执行比对、过滤、条形码计数和UMI计数。它使用Chromium细胞条形码生成特征-条形码矩阵,确定聚类,并进行基因表达分析。Cellranger 计数流程可以从同一 GEM 孔上的多次测序运行中获取输入数据。该流程可以分析 Chromium 3′ 基因表达检测(例如:3’ v1、v2、v3、5’ v1),但不支持较新的数据类型(例如,3’/5’ 样本多重化、Flex 或 5’ 抗原捕获)。cellranger vdj该软件读取V(D)J文库的FASTQ文件,并进行序列组装和配对克隆型鉴定。它利用Chromium细胞条形码和UMI对每个细胞的V(D)J转录本进行组装。克隆型和CDR3序列以.vloupe文件的形式输出,该文件可加载到Loupe V(D)J浏览器中。cellranger aggr汇总多次运行 CellRanger Count、CellRanger VDJ 或 CellRanger Multi 的输出结果,将这些运行结果归一化到相同的测序深度,然后重新计算特征条形码矩阵并对合并后的数据进行分析。cellranger reanalyze它获取由 cellranger count、cellranger multi 或 cellranger aggr 生成的特征条形码矩阵,并使用可调参数设置重新运行降维、聚类和基因表达算法。cellranger annote它接受 molecule_info.h5 和 cellranger count、cellranger multi 或 cellranger aggr 生成的可选 .cloupe 文件,以生成细胞类型注释。
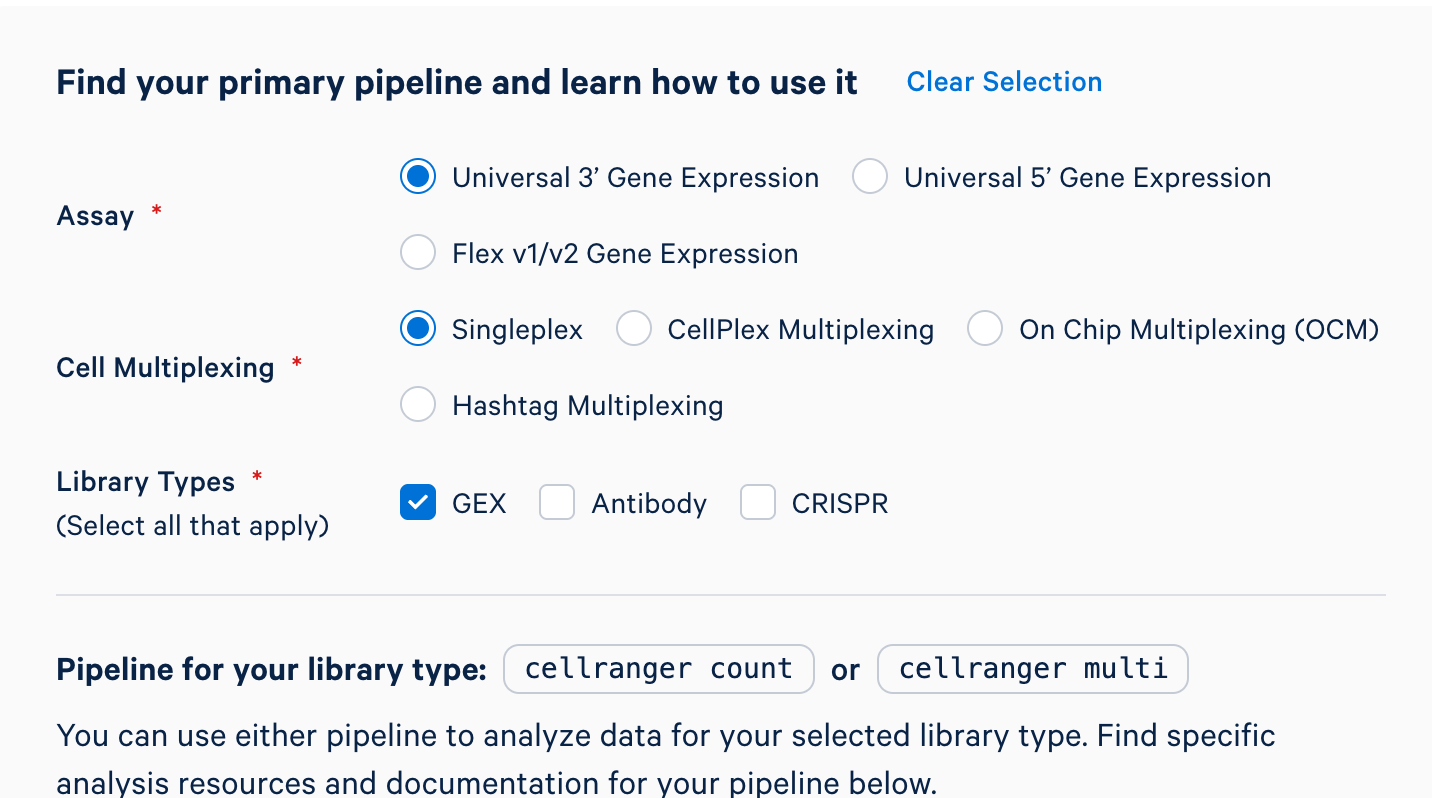
4.1 选择Pipeline
10x Genomic官网有推荐的pipline选择,直接选择不同的平台即可出现管线
4.3 修改文件名称
cellranger的输入文件格式是fq格式,并且文件的命名也是有要求,文件命名格式如下:
[Sample Name]_S1_L00[Lane Number]_[Read Type]_001.fastq.gz
- I1:sample index read(可选)
- R1:Read1 26bp UMI
- R2:Read2 测序数据
ls *.gz|cut -d "_" -f 1|uniq|while read id; do mv ${id}_1.fastq.gz ${id}_S1_L001_R1_001.fastq.gz; mv ${id}_2.fastq.gz ${id}_S1_L001_R2_001.fastq.gz;done4.4 执行cellranger count
修改好文件之后,进行count命令
STAR 比对需要大内存,为了内存不吃紧,出现 chunk 失败,样本循环处理,将cellranger标准代码扔给AI,写一个循环。等前一个样本跑完,再开始下一个。这样能避免机器被一次性跑爆内存/CPU。
#!/bin/bash
# 设置参考基因组路径
ref=/home/erwin/projectQiao/PRJNA614539/1cellranger/reference/refdata-gex-GRCh38-2024-A
# fastq 路径
FASTQ_DIR=/home/erwin/projectQiao/PRJNA614539/SRRdata/data
# 日志目录
LOG_DIR=/home/erwin/projectQiao/PRJNA614539/0cellranger_logs
mkdir -p $LOG_DIR
# 遍历所有样本
for id in $(ls $FASTQ_DIR/*_S1_L001_R1_001.fastq.gz | xargs -n1 basename | cut -d "_" -f 1 | sort | uniq); do
echo ">>> 开始处理样本: $id"
LOG_FILE="$LOG_DIR/${id}.log"
cellranger count \
--id=$id \
--transcriptome=$ref \
--fastqs=$FASTQ_DIR \
--sample=$id \
--nosecondary \
--localcores=90 \
--create-bam=true \
> $LOG_FILE 2>&1
echo ">>> 样本 $id 完成,日志: $LOG_FILE"
done
echo ">>> 所有样本运行结束!"cellranger count 运算还是很慢的,需要比较长时间
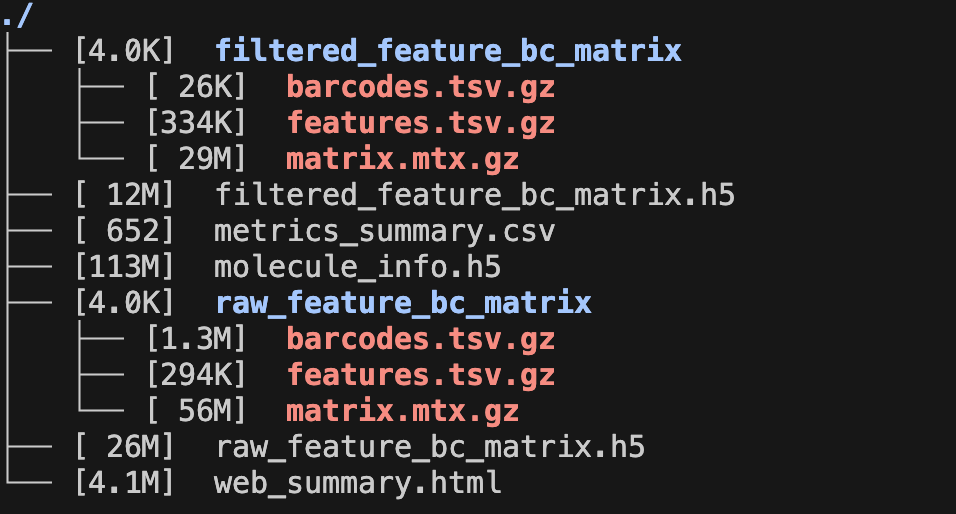
5. CellRnager Count结果解读
主要的结果文件都在out 文件夹里面
- filtered_feature_matrix
MEX格式 表达矩阵(三个文件组合) - barcode.tsv.gz 细胞条形码 - feature.tsv.gz 基因信息 - matrix.mtx.gz 表达数据
- filtered_feature_bc_matrix.h5
- raw_feature_bc_matrix 原始数据
- web_summary.html 网页版的质控报告
- metric_summary.csv 质控指标的表格版
5.1 细胞和基因数目的评估
对样本中的细胞和表达的基因个数评估,同时还给出了barcode,index,umi,RNA reads不同的序列Q30
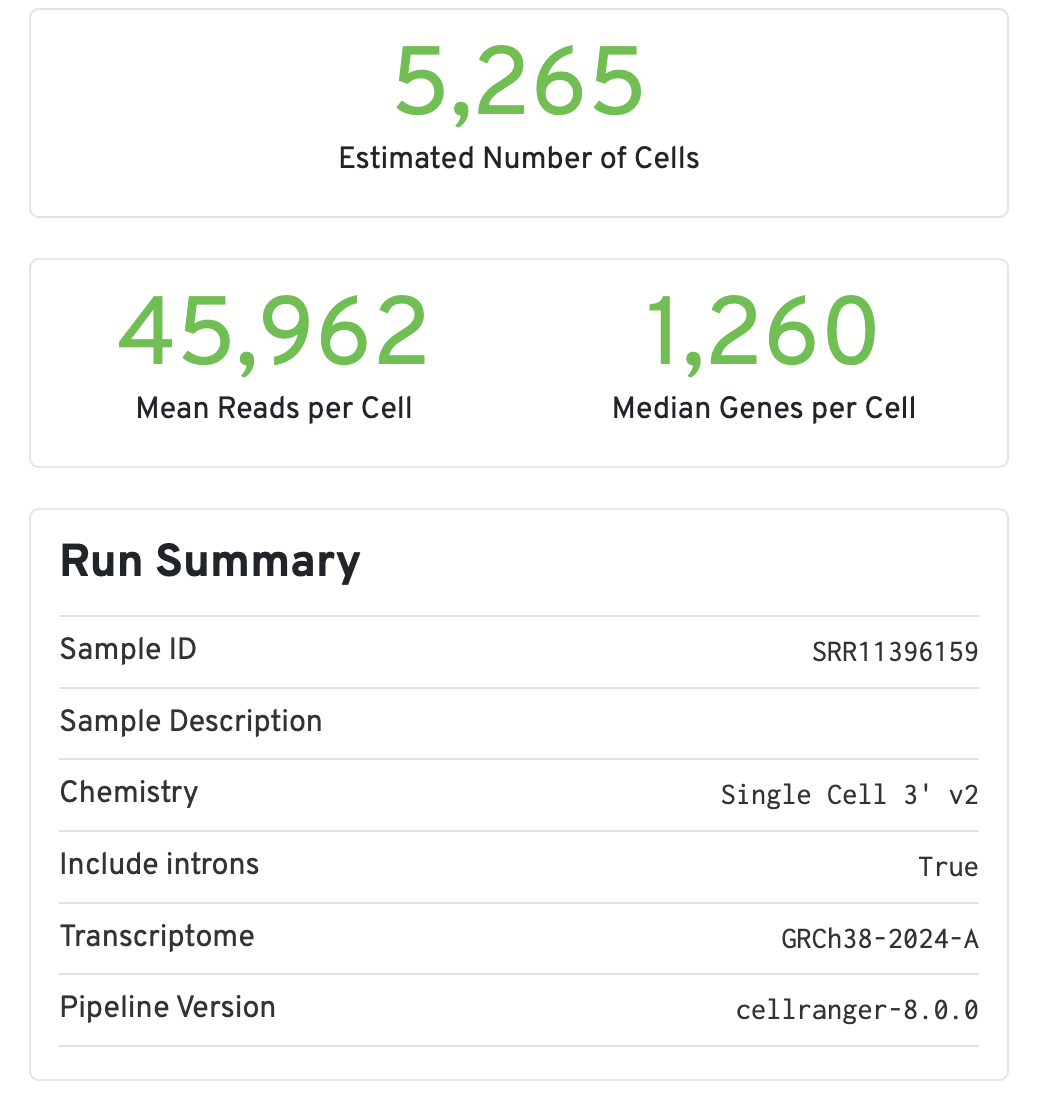
- Estimated Number of Cells 估计细胞数,捕获效率良好
- Median Genes per Cell 中位基因数,细胞质量高 >1000
- Mean Reads per Cell 中位UMI数 测序深度充足 >5000
Sequencing 数据框内内容 - Sequencing Saturation 测序饱和度 50-80% - Q30 Bases in Barcode 数据质量
Mapping数据内容
- Reads Mapped to Genome 94.8%
- Reads Mapped Confidently to Genome 91.6%
- Reads Mapped Confidently to Intergenic Regions 3.6%
- Reads Mapped Confidently to Intronic Regions 27.4%
- Reads Mapped Confidently to Exonic Regions 60.5%
- Reads Mapped Confidently to Transcriptome 75.7%
- Reads Mapped Antisense to Gene