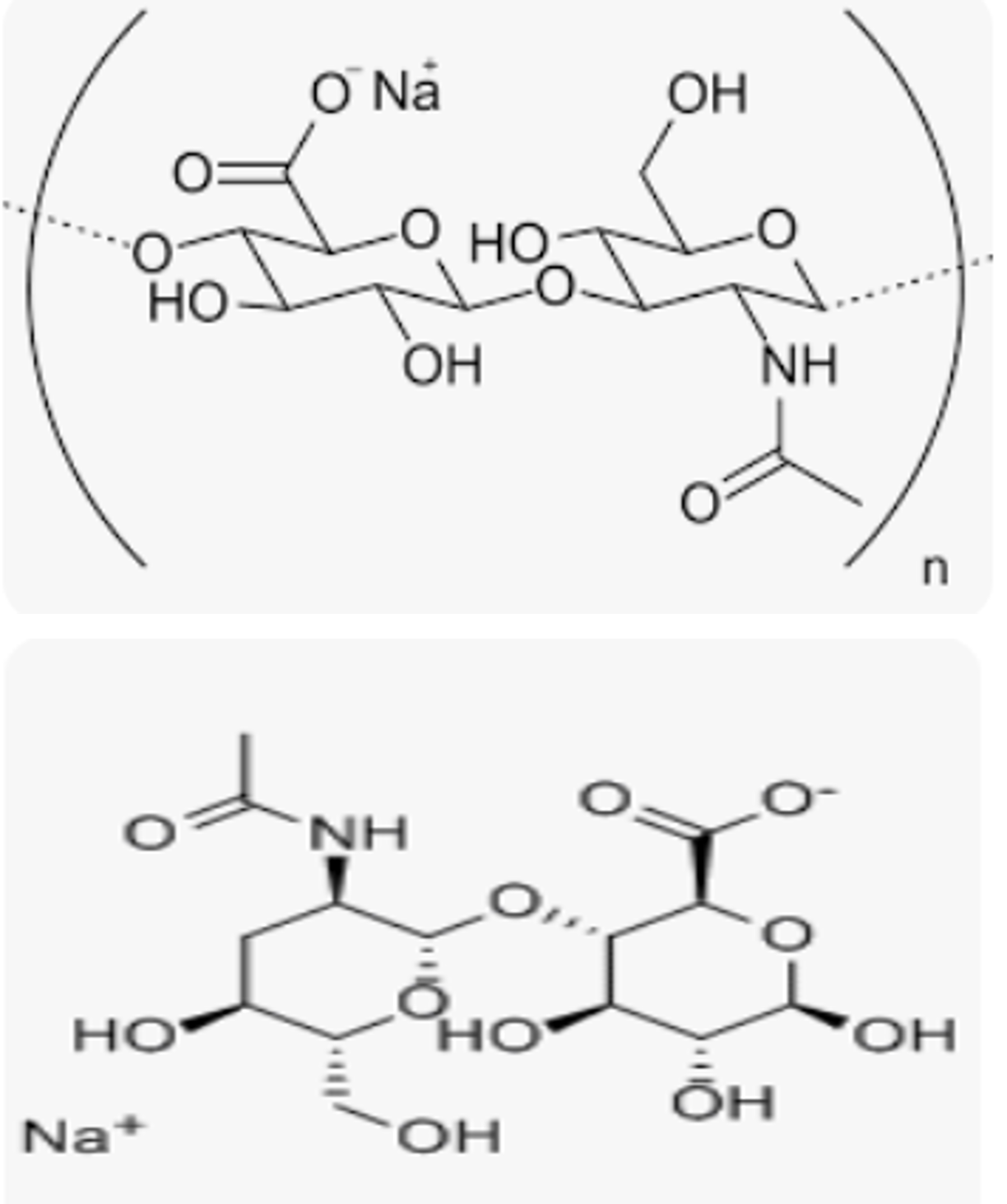
Abstract
肠道内微生物群落的组成可能会影响大脑、肺和皮肤等远处器官的功能。这些微生物可以促进疾病,也可以发挥有益作用,这引发了肠道微生物可以解释肠道疾病和皮肤疾病同时发生的假设。皮肤创伤导致真皮层破坏或真皮透明质酸降解,会导致宿主防御基因 Reg3 和 Muc2 在结肠中的表达增加,并且皮肤创伤会改变肠道细菌的组成和行为。通过暴露于这些皮肤干预所释放的透明质酸,体外可诱导 Reg3 和 Muc2 表达增强。皮肤受伤后结肠微生物群的变化具有重要的功能性,因为这些细菌能够穿透肠道上皮,增强葡聚糖硫酸钠 (DSS) 引起的结肠炎,这一点可以通过在无菌小鼠中使用口服抗生素来挽救皮肤相关的 DSS 结肠炎,以及将皮肤受伤小鼠的粪便微生物群移植到未受伤的小鼠身上来证明。这些观察结果表明皮肤损伤会破坏肠道宿主防御的体内平衡并改变肠道微生物群,从而为皮肤-肠道轴的存在提供了直接证据。
Intruduction
这些上皮表面的疾病或损伤会导致物理和免疫屏障被破坏,从而改变驻留微生物的免疫稳态,并可能使某些生物体促进疾病[Levy et al. (2017)](Belkaid and Hand 2014)。多种皮肤和肠道疾病经常同时发生,例如特应性皮炎和食物过敏,或者银屑病和炎症性肠病(IBD)[Kim et al. (2017)](Weidinger and Novak 2016)。这些临床观察表明肠道和皮肤之间存在功能轴。肠道、肺和大脑之间也发现了类似的器官间通讯[Dickson et al. (2016)](Agirman, Yu, and Hsiao 2021)。
研究表明,小鼠皮肤受伤会导致口服葡聚糖硫酸钠 (DSS) 后对结肠炎的敏感性增加,或在 IL10−/− 小鼠的自发性结肠炎实验模型中(Dokoshi et al. 2021). 在一种基因小鼠模型中也观察到了类似但更严重的表型,该模型通过在表皮中靶向转基因过度表达人类透明质酸酶-1(HYAL1)来促进皮肤中透明质酸(HA)的讲解。
在本研究中,我们对皮肤受伤后的肠道和粪便微生物组或表皮中透明质酸酶的表达进行了详细分析,以模拟组织损伤的这一方面。肠道中的这些变化通过实验证明皮肤影响结肠中的基因表达,从而提供了皮肤至肠道免疫轴的证据。
Results
皮肤中的透明质酸酶活性诱导结肠中的 Muc2 和 Reg3 表达
为了更好的理解皮肤是怎样可能影响肠细胞基因表达,单细胞测序scSeq对整个对皮肤干预的小鼠结肠(the whole colon)进行测序,通过角蛋白14(K14)启动子的条件性调控,实现透明质酸酶1(HYAL1)的转基因表达。这个模型概述了没有导致免疫细胞迁移或者系统细胞因子反应的皮肤受损,并且模型已经展示了口服葡聚糖硫酸钠(DSS)后,患结肠炎的可能性大大增加。导致结肠疾病易感性增加的机制尚不清楚,并被用作人类皮肤炎症和 IBD 之间关联的模型。
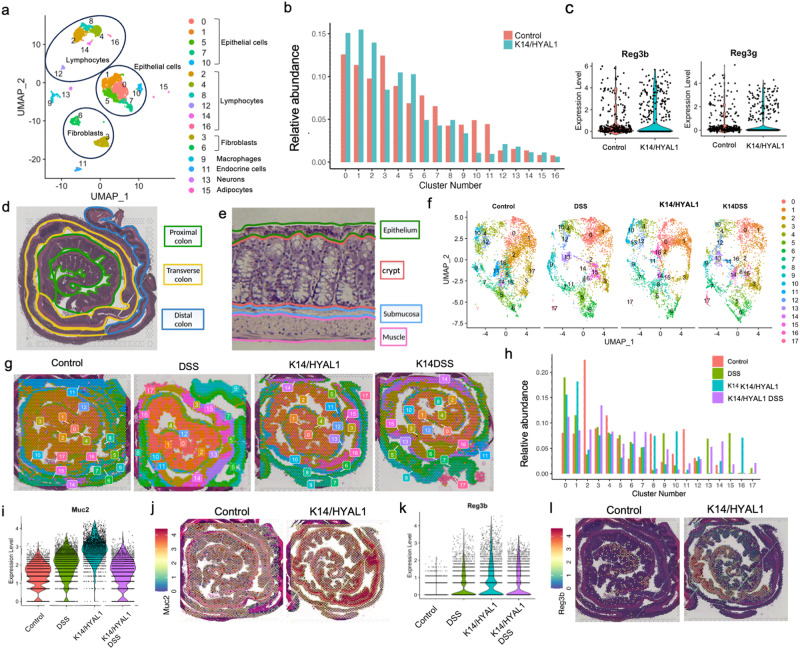
scSeq 数据分析分辨出了 17 个不同的细胞簇,其中包括上皮细胞和淋巴细胞簇,在 K14/HYAL1 小鼠中有所增加(Figure 1a)。重要的是抗微生物和宿主反应的基因 Reg3b, Reg3g, Muc2, and anterior gradient 2 (Agr2)是增加的在肠上皮细胞中。Figure1c中展示了Reg3b和Reg3g基因。为了验证之前的发现,空间转录组用来研究盲肠卷到直肠后,对整个结肠进行肠道分析,以便对结肠的不同区域进行全长分析(Figure 1d,e). 这些数据被分为18种簇,并对这些簇进行映射,区分位于近端、横结肠和远端结肠的转录本。Several clusters were differentially expressed between experimental groups of K14/HYAL mice compared to control, with greatest enrichment of clusters 1 (Epithelium), 10 (Epithelium) and 16(Crypt)(Figure 1h). 有趣的是,即使 DSS 在肠道中引起大量炎症反应,K14/HYAL1 小鼠在没有 DSS 刺激的情况下仍表现出最高的 Muc2 表达水平。还有Reg3b和Reg3g的情况同样如此。
基于cluster分类的基因,每个cluster选取3个标志基因,然后进行GO分析,在 K14/HYAL1 小鼠的结肠中检测到了 0-2、4、5 簇的宿主防御反应(Figure S1b,c)。Muc2基因和Agr2基因的小提亲图表达水平可以观察到。
Mucin Production
与对照组相比,K14/HYAL1 小鼠和有皮肤损伤的小鼠的横结肠隐窝中黏蛋白染色均增加(Figure 2a). Western blot 检测显示横结肠中 Reg3 蛋白表达增加,免疫染色进一步表明,无论是 K14/HYAL1 小鼠还是皮肤受伤,Muc2 和 Reg3g 的表达都会增加(Figure 2b,c,d,e)。这一结果表明,皮肤诱导肠道宿主防御基因改变的能力并不依赖于肠道中细菌的存在。
TBP(TATA结合蛋白; TATA-Binding Protein)是一种常用的内参基因,广泛用于基因表达分析中
- 转录因子:TBP是转录因子IID的核心组成部分,负责核心启动子结合,定位RNNA聚合酶II,启动转录过程
- 稳定性
- 广泛表达
Hyaluronan fragments induce expression of Muc2 and Reg3 in colon epithelial cells
由于 K14/HYAL 小鼠全身暴露于 HA 片段的量增加,我们接下来询问这些片段是否可以直接促进培养中 Muc2 和 Reg3 表达的增加。透明质酸片段通过添加到小鼠结肠组织的体外培养物和人类结肠上皮细胞系(HT29)中直接进行测试.两种系统均表明,通过添加透明质酸片段(6.8 kDa HA 或 LMWHA:低分子量 HA)可直接诱导 Reg3 表达(Figure 2g-i).
Skin injury changes the composition and behavior of the gut microbiome
To address this, shotgun metagenomic DNA sequencing was performed on co-housed littermates with and without skin wounding (n = 32 in each group).对照组服用口服万古霉素,这是一种仅限于肠道内作用的不可吸收抗生素。该实验组用于比较皮肤受伤后肠道菌群的变化与这种广谱抗生素引起的肠道菌群的巨大变化.尽管万古霉素诱导的肠道菌群变化最大,但在对照组小鼠和皮肤伤口 2 天后的小鼠中,Shannon α 多样性和 Robust Aitchison β 多样性也存在显著差异(Figure 3a,b). The relative abundance of several bacterial species including Lachnospiraceae bacterium A4 and Akkermansia muciniphila was increased in mice with skin wounds(Fig 3c,d). 皮肤损伤与肠道菌群中与胆碱分解代谢过程和钴胺酸合酶活性相关的基因存在增加有关,这些过程会影响细菌的存活和机会性病原体(如多形拟杆菌)的活性.除了与病原体相关的基因增加之外,皮肤损伤还会导致对肠道有益的基因减少,例如有助于抑制肠道炎症反应的丙酸分解代谢过程.
Methods
Animals and animal care
传统过表达人透明质酸合成酶(C57BL/6背景)的转基因鼠使用之前实验室方法构建(Muto et al. 2014),将组成型启动子和loxP-floxed GFP基因上游是透明质酸酶-1(Hyal1)结合起来。K14-cre 转基因小鼠与 Hyal1 小鼠杂交,产生 K14-cre Hyal1 小鼠。
Cre-lox系统是以小鼠作为研究工具,可对体内进行组织时空特异性的基因敲除、敲入及示踪。
用于与 K14-cre Hyal1 小鼠进行比较的对照小鼠是 Cre 阴性且 Hyal1 杂合的同窝小鼠。无菌小鼠被饲养并安置在柔性薄膜隔离器中直至 6-8 周龄。
Mouse model of DSS colitis
野生型小鼠(C57BL/6) 或者K14-Cre-Hyal1小鼠引用2.5%DSS 7天喂养并且每天测量体重。DSS 给药后第 14 天处死小鼠(无治疗:n = 5,DSS 治疗:对照 n = 8,K14/HYAL1 n = 13)。体重减轻超过原始体重 20% 的小鼠将被实施安乐死。皮肤伤口小鼠或 SPF 组(n = 3)、无菌组(n = 6)及粪便微生物组移植组(n = 8)的对照野生型小鼠,在饮用水中添加 2.5% DSS,持续 5 天后处死。按照下面的指标进行评分
- 隐窝结构(正常,0 - 严重的隐窝扭曲,整个隐窝丢失,3)
- 炎症细胞浸润程度 (正常,0 - 致密炎性浸润,3)
- 肌肉增厚(隐窝底部位于粘膜肌层,0 - 出现明显的肌肉增厚,3)
- 杯状细胞耗竭(缺失,0-存在,1)隐窝脓肿(无,0-有,1)
隐窝是动物学中用于描述动物体内器官或组织结构内凹陷的术语,常见于消化系统、呼吸系统等解剖结构中。其形态和功能与所在器官密切相关,可能承担物质交换、分泌物储存或免疫保护等作用
杯状细胞(goblet cell)是一种能分泌胶状黏蛋白的单层柱状上皮细胞,在气管内,负责分泌使气管湿润的黏液;在肠道内,则负责润滑食糜,使其更容易通过肠道。
硫酸葡糖钠盐(葡聚糖硫酸钠),英文Dextran Sulfate Sodium Salt,缩写DSS,是葡聚糖的聚阴离子衍生物,由葡聚糖和氯磺酸的酯化反应形成。其中含硫量约为17%,相当于葡聚糖分子的每个葡萄糖残糖中平均含1.9个硫酸基团, 主要是采用不同分子量(36000-50000Da)。
炎症性肠炎(IBD)是一种慢性、易反复的胃肠道感染,会提升肠道肿瘤发生的危险性,主要包括UC和克罗恩病(Crohn disease, CD)。自1985年首次报道采用葡聚糖硫酸钠(dextran sulphate sodium, DSS)制备出仓鼠溃疡性结肠炎模型以来,已有大量研究证明DSS结肠炎模型与人类溃疡性结肠炎相似。DSS结肠炎模型的组织学特点、临床表现、发病部位和细胞因子增殖情况都与人类溃疡性结肠炎(ulcerative colitis, UC)极为相似。
Mouse model of skin wounding
在异氟烷麻醉下,在性别和年龄匹配(8-12 周)的成年野生型小鼠背部形成 1.5 厘米全层切口伤口。伤口处于开放状态没有缝合。8小时后,连续5天,给小鼠提供添加2.5% DSS的饮用水。在无菌设施内,采用外科无菌技术处理小鼠。
Mouse colon ex vivo model
从 C57BL/6 小鼠中切除整个小鼠结肠,最初用 5 ml 无菌 DMEM 培养基冲洗,其中补充有 10% (v/v) FBS 和 1% 抗生素溶液 (抗生素抗真菌剂 (100X))。治疗期间,切除并冲洗干净的结肠,在填充DMEM培养基或添加5 μg/ml HA的DMEM培养基(n= 6)后,两端用无菌缝线缝合。将结肠置于37°C、5% CO2的湿化培养箱中孵育15分钟,然后评估对HA的反应。
Intestinal permeability assay
在对血浆中的 FITC 荧光进行荧光分析之前 3 小时,用 FITC-葡聚糖 (4 kDa, Sigma Aldrich) 灌胃
Bacterial culture and count
将粪便样本摇匀并溶于含有 10% 甘油的无菌 PBS 中。(1 g 粪便溶于 2 ml 溶液中)。使用 LIVE/DEAD BacLight 细菌活力试剂盒(Thermo Fisher)对细菌进行染色,并根据制造商的协议使用 BD FACSCanto RUO 机器通过流式细胞仪进行计数,并使用 FlowJo V10 软件进行分析。 (n: Control = 11, Skin wound = 11, K14/HYAL1 = 6, Vancomycin = 5)
Fecal microbiome transplant (FMT)
从对照小鼠或皮肤伤口小鼠中收集小鼠粪便,并储存在含有 10% 甘油的无菌 PBS 中。(1 g 粪便溶于 2 ml 溶液中)。对于 FMT 制备,将样品混合并通过离心机(500 x g,5 分钟)消除颗粒,然后将等分试样储存在 -80°C 下。将等分试样稀释五倍,并以200 μl/只小鼠的剂量进行管饲,持续6天,直至DSS激发结束。实验期间每天都进行管饲。(n= 8)。
Human skin sample collection and IHC
Carnoy’s fluid(卡诺液) 是一种常用的组织固定液,主要用于组织学和分子生物学样本的快速固定,特别适合保存核酸(DNA、RNA)完整性,同时固定细胞和组织结构。
- 对于革兰氏染色,用 Carnoy 液固定组织并嵌入 OCT 化合物中,切成 4um 薄片,并用革兰氏染色剂染色
- 对于过碘酸-希夫 (PAS) 染色,将组织用卡诺氏液固定并嵌入石蜡中,然后用 PAS 染色
- 用于 TUNEL 染色(检测细胞凋亡的一种方法)时,使用了 TUNEL Andy Fluo 594 凋亡检测试剂盒
- HABP,Muc2,Reg3g 染色
In situ hybridization
获取新鲜冷冻结肠组织切片,并在 Carnoy 液中固定过夜。使用 RNAscope 荧光多重分析(Advanced Cell Diagnostics Bio)根据制造商的方案,使用预先设计的探针对切片进行 16 S 染色
空间转录组
将未经处理和经 DSS 处理的 WT 和 K14/HYAL1 小鼠的结肠组织从脂肪组织中清除并纵向切割;用冷磷酸盐缓冲液 (PBS) 清洗以去除管腔内容物。从最近端的部分(即盲肠)开始,将管腔侧朝上,将结肠卷起来,形成近端结肠位于中心、远端结肠位于外部的卷。将卷放入组织学塑料盒中,并在液氮冷却的异戊烷浴中快速冷冻 1 分钟,使用预冷的低温恒温器将 OCT 块切割成 10 微米厚,并将切片转移到 Visium 10x 基因组载玻片上,以适合 6.5 mm2 寡核苷酸条形码捕获区域。将含有结肠组织的实验玻片固定,用苏木精-伊红 (H&E) 染色,并使用 Keyence BZX-700 荧光显微镜 (Keyence) 在 2 倍放大倍数下进行成像。
Data analysis
For mouse colon, the 10X Genomics Cell Ranger version 7.0.1 and Space Ranger version 2.0.1 software pipeline with default parameters were used to perform sample demultiplexing, barcode processing, alignment to the mm10 reference genome, and single-cell gene counting.
Flow cytometry analyses
将从对照组或 DSS 治疗组小鼠的结肠切成小块,然后用 2.5 mg/mL 胶原酶 D 和 30 ng/mL DNAse1 在 37°C 下消化 40 分钟,然后通过 70 µm 过滤器过滤,生成单细胞悬浮液用于 FACS 分析。
- FITC -CD45
- PECy7-CD11b
- FITC-Ly6G
- PE-F4/80
- APC-CD11C
- AF700-MHCII
- APC-eFluro-CD4
- PE -CD19
Microbiome analysis
Bacterial DNA for shotgun metagenomic sequencing was extracted by the QIAamp DNA Stool Mini Kit (QIAGEN). Shotgun metagenomic sequencing was done at the UC San Diego IGM Genomics Center utilizing an Illumina NovaSeq 6000.
- 16S/18S/ITS 扩增测序 → 用于鉴定物种
- 宏基因组 shotgun 测序 → 全基因组无偏测序,可分析物种 + 功能
我们使用 Woltka 流程(pipeline)通过 Qiita 平台来处理宏基因组学数据。This pipeline includes adapter and host filtering with qp-fastp-minimap2, sequence alignment against the Web of Life database using Bowtie2,and resultant taxonomic and functional classification with Woltka. The per-genome BIOM table was used to perform analysis on the observed operational genomic units (OGUs).
Community (alpha and beta) analyses were performed in Python. For alpha diversity analysis, the taxonomic feature table was rarefied to the lowest sequencing depth and calculated using the Shannon Index via Python’s scikit-bio package for alpha diversity.