Cre-Lox
简单、重组率高的Cre-Lox系统
利用Cre-Lox系统,可以在特定细胞、组织或生物体,在特定时间点敲除或表达某个基因。实现对特定基因的时空特异性操作
Cre-Lox系统介绍
Cre重组酶
1981年,Nat Sternberg等报道了一种克隆自大肠杆菌P1噬菌体的蛋白——由343个氨基酸编码,38kDa的 Cre(Cyclization Recombination Enzyme)重组酶,属于酪氨酸重组酶家族,它能特异识别loxP位点,并介导loxP位点间的序列删除或重组。
酪氨酸重组酶类在活性位点上有一个Tyr残基,其侧链攻击连接到DNA,使得DNA之间能够交叉重组。该酶能特异性识别并催化34bp的loxP位点间的DNA发生位点特异性重组,反应无需能量辅助因子且快速达到动态平衡
Lox位点
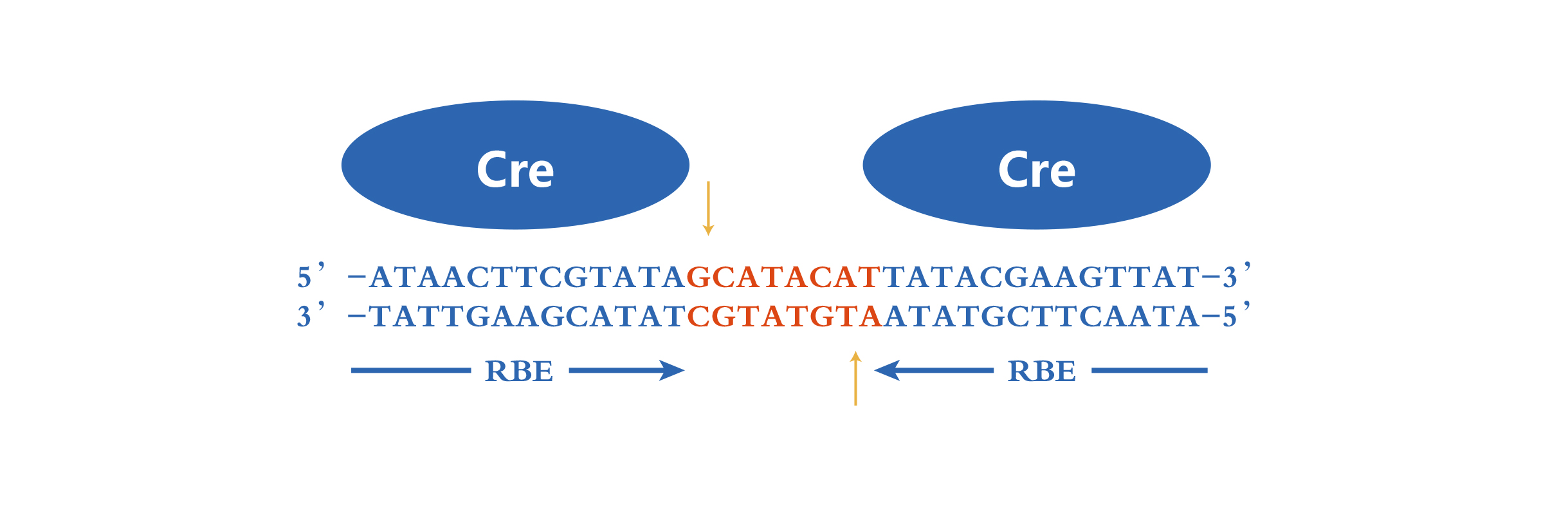
Cre重组酶识别的回文DNA位点,也叫loxP (locus of X-over P1) 位点,长 34bp,其特征结构为ATAACTTCGTATA -NNNTANNN-TATACGAAGTTAT,方向是5‘-3’。两边反向互补的13个碱基为Cre重组酶的识别序列,中间的8个碱基为重组发生位置,这也决定了loxP的方向。N表示可变碱基,不同的碱基选择可形成不同的 Lox位点。
下图是LoxP位点及其突变体位点,解决基因泄露的问题
ATAACTTCGTATA - GCATACAT - TATACGAAGTTAT
\______/ ||||| \______/
左臂(13bp) 间隔区 右臂(13bp)
- 13bp重复序列:是 Cre酶的识别位点,两个重复序列是完全相同的
- 8bp中间间隔序列:不对称,决定了 loxP 的方向性;重组的结果(如删除 vs 翻转)取决于两个loxP的方向。
当基因组内存在loxP位点时,一旦有Cre重组酶,便会结合到loxP位点两端的反向重复序列区形成二聚体。此二聚体与其他loxP位点的二聚体结合,进而形成四聚体。随后,loxP位点之间的DNA被Cre重组酶切下,切口在DNA连接酶的作用下重新连接。
loxP 位点的方向是由其 中间的 8 bp 非对称间隔区(spacer)序列 决定的
- 切除:当两个Lox位点在同一染色体上且方向相同时,将切除同向Lox位点之间的DNA序列(也叫Lox侧翼序列,Flox序列)。
- 反转:当两个Lox位点位于同一染色体上且方向相反时,两个Lox位点之间的序列发生序列反转,即颠倒。
- 易位:如果两个Lox位点位于不同的染色体上且方向相同,则易位事件将导致DNA片段的交换。
- A loxP位点的基本结构
- B Cre-loxP介导的易位重组
- C Cre介导两同向loxP位点间的切除重组
体内Cre-lox系统的基础应用
利用Cre-Lox系统在小鼠体内实现对基因的时空特异性打靶(表达或敲除),原则上需要建立两种小鼠。
首先,建立特异性Cre小鼠,该小鼠中的Cre重组酶由特定启动子驱动,可在特定细胞或组织或全身表达Cre重组酶。Cre重组的特异性由驱动Cre的启动子或增强子控制。
其次,需要建立Flox小鼠,即在该小鼠靶基因序列侧翼带有Lox位点。将上述两种小鼠杂交繁育,子代中可以获得既带有Cre又带有Flox基因的小鼠,即条件性基因打靶(表达或敲除靶基因)小鼠。
“Flox小鼠”是指基因中特定位点被两个loxP序列夹住(flanked by loxP)的小鼠,即带有“floxed基因”的转基因小鼠。
条件性基因表达
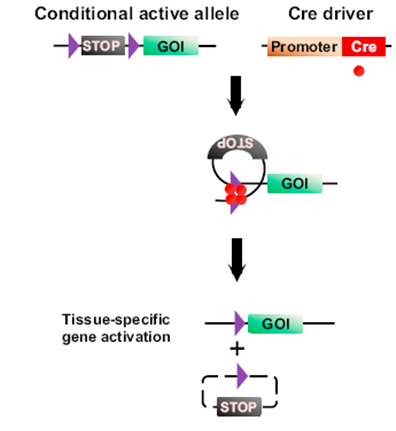
条件性基因表达所需要的Flox小鼠,一般带有一个沉默的转基因,也就是说在靶基因与启动子之间有一个“终止盒”(lox-stop-lox)。这个“终止盒”通常由强polyA信号和/或剪接供体序列构成,能阻止转基因的转录。这些Flox小鼠与细胞类型特异性表达Cre的转基因小鼠交配后获得的子代小鼠中,在每个表达Cre重组酶的细胞中,终止盒被Cre切除,因此仅在这些细胞中表达所需的转基因。
同向切除原理
条件性基因敲除
条件性基因敲除所需要的Flox小鼠,一般在需要切除的DNA片段两侧带有同方向的两个Lox位点(lox-GENE-lox)。与细胞类型特异性Cre小鼠交配后,Cre 重组酶会识别Lox位点,导致靶基因被敲除。由于 Cre 基因的表达受上游启动子的调控,因此启动子的时空特异性就决定了基因重组的时空特异性。
下图展示了基因敲除的原理
Cre-Lox系统迭代
更加准确的进行遗传功能研究,时间特异性的Cre-Lox,即
传统的Cre-Lox本身做不到时间特异性
- 在目标基因的上下游插入两个LoxP位点(flox基因)
- 用一个特定的启动子驱动Cre重组酶基因的表达
- 启动子只要一活跃,Cre就会持续表达–> 立刻切除或倒位两个LoxP之间的DNA片段
- 如果启动子是全身且胚胎(CMV,CAG启动子)
- 如果用组织特异性启动子(Albumin启动子在肝脏,GFAP启动子在星形胶质细胞),只能做到空间特异性
CreER系统
他莫昔芬(Tam)诱导的Cre系统–CreERT2在体内对药物诱导的敏感性更高
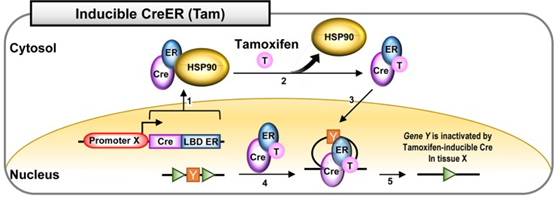
CreERT 是一种融合蛋白,由人雌激素受体(ER)的突变配体结合域与 Cre 重组酶组成。它的活性可以由 4-羟基-他莫昔芬(OHT)诱导,但不受天然 ER 配体的影响。
- 在没有他莫昔芬的情况下,CreER融合蛋白与热休克蛋白90(HSP90)相互作用并存在于细胞质中
- 给予他莫昔芬药物处理会破坏HSP90与CreER的相互作用
- ER与Tam的相互作用诱导Cre的核易位
- 在细胞核中,CreER识别loxP位点(图5.4)并使组织X中的基因Y失活
上图简略表示CreER系统步骤
- 核中在PromoterX启动子催动下表达Cre/LEB ER蛋白,形成Cre/ER/HSP90复合体
- 加入Tamoxifen后,HSP90解离开,剩下Cre/ER/T复合体
- Cre复合体作用在Loxp上,进行基因编辑
Cre/Tet系统
四环素诱导系统(tetracycline)或其衍生物 doxycycline(多西环素)
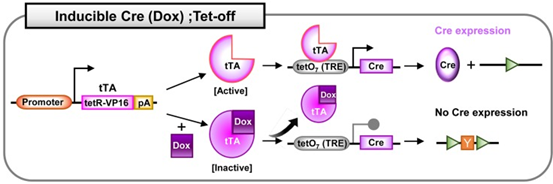
四环素诱导系统,也叫强力霉素(Dox,四环素衍生物)诱导的Cre系统。该系统有两种模式:Tet-on和Tet-off,分别是Dox依赖性Cre的激活和Dox依赖性Cre的失活。
顺式作用元件(cis-regulatory element,CRE),是与结构基因串联的一段非编码DNA序列,一般位于转录点上游,作用是调节邻近基因的转录。
反式作用因子(英语:Trans-acting factor),也称为反式因子,是指能直接或间接地识别或结合在各类顺式作用元件核心序列上,参与靶基因转录效率调控的蛋白质或RNA。
Cre/Tet-on系统
核心系统:
- 反向四环素控制反式激活因子(reverse tetracycline-controlled transactivator,简称 rtTA)
- 四环素响应元件(TRE),通常是19个核苷酸的四环素操纵子(tetO)序列的7个重复,调节Cre基因的表达。
在Tet-on系统中,表达普遍存在的或组织特异性的启动子驱动的rtTA。在没有Dox的情况下,失活的rtTA无法与负责调控Cre基因的TRE序列结合,则Cre不表达。在Dox给药之后,Dox结合并激活rtTA。激活的rtTA与TRE序列结合并诱导Cre表达
Cre/Tet-off系统
核心系统:
- 四环素控制反式激活因子(tetracycline-controlled transactivator,简称 tTA)
- 四环素响应元件(TRE),通常是19个核苷酸的四环素操纵子(tetO)序列的7个重复,调节Cre基因的表达。
在Tet-off系统中,在没有Dox的情况下,激活的tTA能够结合Cre前的TRE序列并诱导Cre表达。而在Dox给药后,与Dox相互作用的tTA被灭活。灭活的rTA不再与TRE结合,因此Cre表达受到抑制。